DNA-LNP? Eine Spurensuche
Jede Verunreinigung hat eine Messung, von der sie nicht erfasst wird.
In jeder bislang unabhängig untersuchten Pfizer-BioNTech-Ampulle befinden sich neben Billionen modRNA-Molekülen auch Milliarden Fragmente linearisierter Plasmid-DNA; nur möglich mit systematischen Vergehen in Herstellung und Freigabe.
Wie kommt die DNA in die fertige Ampulle? Verfügbare Informationen zur Herstellung werden präsentiert und mit dem Industriestandard verglichen.
Was wussten die Behörden? Relevante Zulassungsunterlagen werden in den Kontext neuester IFG-Antworten der EMA gesetzt.
Wie kann es dazu kommen, dass ein so verunreinigtes Produkt hergestellt und freigegeben wird? Das verantwortliche Ensemble an Analysemethoden wird auf Schwächen untersucht.
Anmerkung: Wenn zu einer Behauptung eine Fußnote fehlt, kommt sie zu einem späteren Punkt im Text. Mehrere Fußnoten auf eine Zahl zu beziehen scheint auf substack nicht möglich, und ich wollte nicht leere Fußnoten einbauen, die nur auf andere Fußnoten verweisen.
Der Herstellungsprozess
Die “Prozess 2”-Wirkstoffherstellung beginnt mit Plasmiden.1 Diese kreisförmigen, synthetisierten DNA-Moleküle enthalten ausser der BNT162b2-Sequenz auch für die bakterielle Vervielfältigung wichtige Elemente.2 Sie werden per unbekannter Methode in genveränderte3 E.coli-Bakterien transfiziert, vermehrt und aufgeteilt, um die Referenz-Zellkultur zu generieren.
Von dieser Referenz werden Arbeits-Zellkulturen gebildet, mit denen Nährstofftanks “inokuliert” werden. Die Bakterien vervielfachen sich exponentiell per Fermentierung; Antibiotikaresistenz-Gene für Neomcyin und Kanamycin stellen sicher, dass nur transfizierte E.coli überleben. Nach Erreichen der gewünschten Konzentration wird die Plasmid-DNA wieder aus den Bakterien herausgeholt, indem durch Zufuhr einer chemischen Lyse die Zellmembran aufgelöst wird. Diese Plasmid-E.coli-Lyse-Brühe wird mit Ultrafiltrierung/Diafiltrierung und Anionenaustauschchromatographie4 gereinigt. Schliesslich wird die aufbereitete DNA “linearisiert”; sprich per Zugabe eines sogenannten Restriktionsenzyms wird das kreisförmige Plasmid an einer gezielt platzierten Stelle in der Sequenz zu einem Strang aufgeschnitten.
Die Herstellung der linearisierten DNA wird in den EMA-Leak-Dokumenten nur rudimentär beschrieben. Zum Beispiel werden Salze verwendet, um die Aktivität des Restriktionsenzyms bei der Linearisierung zu beenden; diese Salze müssen jedoch entfernt werden, um die später verwendete DNase nicht zu behindern. Kevin McKernan hat bereits im März den Beweis5 erbracht, dass diese Reaktion unvollständig ist, da bei der Sequenzierung auch Reads mit intakter Enzym-Schnittstelle sind.
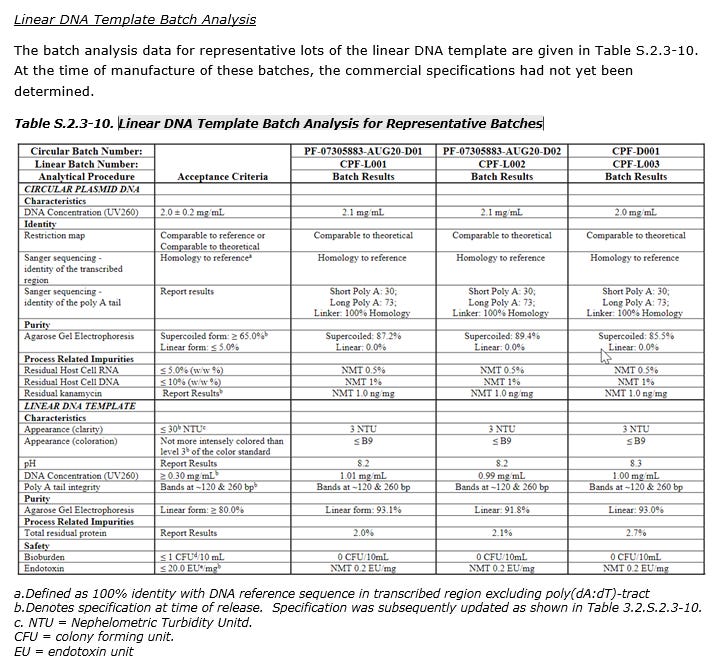
Die Wirtszellen für die Plasmid-DNA sind genetisch modifiziert,6 was um so dringender in Frage stellt, wie effektiv das Ausleiten der Bakterienreste ist. Die Endotoxin-Grenzwerte liegen mit ≤20 EU/mL für die lineare DNA7 und ≤12,5 EU/mL für den Wirkstoff8 über den <10 EU/mL von Vitamin B12 Depotspritzen9 - trotz der chromatographischen Reinigung. Die unterschiedlichen Gleichheitszeichen sind gewollt, denn wie Pfizer in einem umfangreich geschwärzten Antwortschreiben auf einen FDA-Qualitäts-Fragenkatalog von Juli 202110 erklärt, wird Endotoxin als “action limit”-Grenzwert gehandhabt, also wird erst bei Überschreitung eingegriffen. Weswegen der FDA diese vermeintlichen typographischen Fehler11 erst sieben Monate nach Markteinführung auffallen sei der Spekulation überlassen, ebenso Pfizer’s Kriterien für die Auswahl der Grenzwerte mit “action limit”.
Wie später im Kontext der Wirkstoffherstellung näher erläutert, hat die Reinigungsmethode Ultrafiltrierung/ Diafiltrierung (UFDF) bei Nukleinsäure-Enzym-Produkten industrieweit seit Jahrzehnten anerkannte Schwächen.12 Die Haftungsbefreiung entfernt jedoch den finanziellen Anreiz, ein teureres Produkt herzustellen.
Dieser Prozess-Abschnitt findet am Pfizer-Standort Chesterfield statt, inklusive Züchtung der “Master Cell Bank” oder Referenz-Zellkultur am 20.04.2020.13
Prozess 1, womit sämtliche für die Erstzulassung relevante Daten erzeugt wurden, arbeitet mit PCR-kopierten DNA-Templates - viel höhere Reinheit, viel kleinere Chargengröße.14
Beteiligte Standorte der Wirkstoff-Herstellung sind Pfizer Andover und BioNTech Mainz & Rentschler. Die linearisierte Plasmid-DNA (linear DNA template)15 ist die wichtigste Zutat bei der Herstellung des aktiven Wirkstoffes modRNA durch IVT (in-vitro Transkription).16 Ein 50L-Bioreaktor wird mit den RNA-Bausteinen Cytosin, Adenosin, Guanin und N1-Methylpseudouridin statt Uracil gefüllt. Mit RNase-Inhibitor zum Verhindern von RNA-Denaturierung wird die lineare DNA-”Schablone” eingespeist. Die Reaktion einleitend wird T7 Polymerase zugeführt, um aus der linearen DNA modRNA zu “transkribieren”. Die lineare DNA wird dabei nicht abgebaut. Dabei entstehen Pyrophosphate, die Magnesium binden, welches die T7 Polymerase aber braucht, also wird Pyrophosphatase beigefügt um das gebundene Magnesium durch Zersetzung der Pyrophosphate wieder zu befreien.
Nach einer letzten Inkubationszeit endet die IVT, und die DNase-I-Verdauung wird umgehend eingeleitet. In etwa achtfacher Konzentration verglichen zur Plasmid-DNA wird die DNase untergemischt. Bei diesem Schritt entstehen die DNA-Fragmente, in der Größenordnung von ~100 Milliarden Fragmenten pro späterer Dosis, durch das Zerschneiden der ~7800-Basenpaar DNA-Templates in durchschnittlich ~110 Basenpaar-Fragmente.17 Pfizer-BioNTech-Zulieferer Thermo Fischer18 stellt einen bemerkenswert unbefolgten Ratgeber für DNase zur Verfügung, mit Hinweisen wie etwa Verdünnung der Lösung vor DNase-Zufuhr, mehrere/längere Anwendungen, oder, dass Aktivität gegen RNA:DNA-Hybriden im Vergleich zu DNA auf eine Nachkommastelle eingeschränkt ist.19 Es wird auch darauf hingewiesen, dass DNase sehr klebrig ist und bis zu 50% der zugeführten Menge innerhalb von zehn Minuten an der Gefäßwand kleben kann.20
Die DNase-Reaktion wird nach 29-35 Minuten durch Zugabe von EDTA-Salzen beendet und die Proteinase-K-Verdauung beginnt.21 Dieser Schritt zerkleinert Proteine statt DNA. An Stelle eines definierten Endpunktes wird die Temperatur nach 10- bis 15-minütigem Mischen reduziert und der in Behälter umgefüllte “Proteinase-K-Pool” hat dann 96 Stunden Zeit, den LNP-Formulierungs-Standort22 zu erreichen.
Der “Prozess 1”-Wirkstoff, mit dem die Impfstoffe für die klinischen Studien formuliert wurden, hatte statt der Proteinase-Verdauung ein hocheffizientes und nicht skalierbares Aufbereitungssystem mit Magnetkügelchen - also nicht nur zwei Enzyme weniger23 in der Herstellung, sondern auch eine für die Massenproduktion zu aufwändige Reinigung.
Wie auch bei der Plasmid-DNA kommt Ultra-/Diafiltrierung24 zum Einsatz; hier sollen die DNase- und Proteinase-verdauten “Schnipsel” ausgesiebt werden, während die modRNA dank einer theoretischen Länge von 4283 bp zurückbleibt. Dieser “Konfetti-Ansatz” ist für den letzten Schritt der Wirkstoffherstellung notwendig, welcher in Deutschland am Standort Rentschler nach Transport des Proteinase-Pools aus Mainz durchgeführt wird.
UFDF-Prozedur25 wie folgt: Verdünnung mit fünffachem Volumen an Diafiltrierungs-Pufferlösung, Konzentration der Lösung durch Drucksieben, Verdünnung mit 10-facher Formulierungslösung mit anschließender Aufkonzentration. Der resultierende UFDF-Pool wird abwechselnd Feinfiltrierung und UV-Spektroskopie-Messungen des RNA-Gehalts unterzogen. Nach einer dritten und letzten sterilen Filtration erfolgt die Freigabetestung der Wirkstoffcharge und die einzige Bestimmung des DNA-Gehalts. Im EMA-Leak werden Werte für Restmengen von Proteinase K und T7 Polymerase26 angegeben, aber nicht die zugrundeliegende Analysemethoden, und auch keine Informationen zu den anderen Enzymen. Da die gesamte verbleibende Produktionskette bis hin zur Nadel im Arm keinen Einfluss27 auf den DNA-Gehalt hat, ist sie vorerst unwichtig.
UFDF ist der einzige dedizierte Schritt zur Aufbereitung der modRNA. Artikel von vor zehn Jahren betonen, dass UFDF alleine kein ausreichend reines Produkt erzeugen kann, und dass die filtrierte Lösung weitere Aufbereitungsschritte benötigt.28 Industriebroschüren zeigen, dass wesentlich geringere DNA-Grenzwerte für Impfstoffe bereits bestehen.29 Wie anfangs erwähnt erhält die Plasmid-DNA neben UFDF auch eine zusätzliche Reinigung mit Anionen-Chromatographie. Wieso wurde diese Methode nicht auch für den Wirkstoff angewandt?
Ein japanischer Patentanwalt hat unter dem Pseudonym
eine kolossale Auswertung der verfügbaren Datensätze30 als Kritik an der qPCR-Methode zur Bestimmung von Rest-DNA veröffentlicht, und beinhaltet u.a. eine rigorose Kalkulation des tatsächlichen DNA-Gehalts der verschiedenen gemessenen Produkte auf 79,4-116 ng/Dosis. Auf die Messmethoden werde ich später (und weniger minutiös) eingehen.Der Ausgangspunkt
Das seit Anfang 2021 kursierende EMA-Leak31 enthält genug Information, das mir bis heute neue Sachen auffallen.32 Es ist der einzige gänzlich ungeschwärzte Einblick in die Herstellung. Das älteste “Rapporteur Rolling Review”-Dokument33 ist auf 30.11.2020 datiert, einen Tag vor der Autorisierung unter Regulation 174 in Großbritannien, zehn Tage vor der ersten von unzähligen34 FDA “Emergency Use Authorizations”, und drei Wochen vor der ursprünglichen “Conditional Marketing Authorization” oder CMA seitens der EMA am 21.12.2020.35
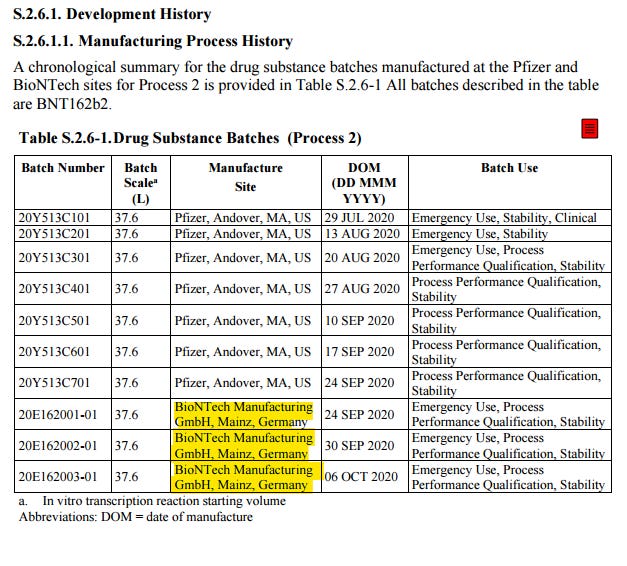
Pfizer-BioNTech’s Antwort auf Frage 14 im Dokument “M1 20 Nov 2020 FDA Query Responses” stellt klar;36 für keins der fünf Enzyme werden nach Anlieferung die Händlerangaben validiert, jedoch verlassen sich die Hersteller auf eben jene Angaben, um die erforderlichen Konzentrationen zu bestimmen.37 Die Marktzulassung erfolgte zunächst nur für den amerikanischen Standort Andover, weil für die beiden deutschen Standorte Mainz & Rentschler noch keine Daten38 eingereicht waren - Wirkstoff hergestellt haben die Standorte trotzdem.39
zeigte, dass BioNTech bereits Q2 2021 Dienstleister für skalierte Plasmid-Fertigung inklusive Fertigung und Aufbewahrung der Plasmidkulturen unter Vertrag genommen hat,40 also ist davon auszugehen, dass auch vor der Einweihung des BioNTech-Plasmidstandortes in Marburg durch Olaf Scholz Anfang 202341 linearisierte DNA nicht nur von Pfizer’s Chesterfield-Anlage hergestellt wurde. Zu BioNTech-Plasmidchargen gibt es meines Wissens noch keinerlei Unterlagen.Die europäische Marktzulassung erfolgte trotz der dokumentierten Mängel, welche als “Spezifische Verpflichtungen” und “Empfehlungen” (Specific Obligations SO42 bzw. Recommendations REC)43 zur weiteren Entwicklung in den ursprünglichen EPARs zu finden sind.44 Obwohl die Enzymthematik mit großen Fragezeichen versehen war, u.a. bewerteten Pfizer-BioNTech die Menge zugeführter Enzyme nicht als kritischen Prozessparameter,45 beziehen sich lediglich Empfehlungen #3 & #7 darauf. Auf der Internetseite der EMA befindet sich eine tabellarische Auflistung aller Änderungen der Zulassung, darunter auch die Erfüllung dieser Auflagen.46 Zu diesen Produktänderungen gibt es falls überhaupt nur einen zensierten CHMP EPAR-Bericht, es sei denn, jemand fragt nach, was bei den Unterlagen zur Enzymaktivität geschehen ist. Sie zeigen, wie unter Ausschluss der Öffentlichkeit das Verhältnis zwischen Pharma und Staat immer dysfunktioneller wurde.
Das große Mauern
Dank einer IFG-Anfrage an die EMA zu den DNA-Verunreinigungen47 gibt es Einblick in vier Dokumente, herausgegeben am 14.11. und 6.12.2023.
Assessment report for the Post-Authorization Measure REC 027
Einreichung: 30.03.2021
CHMP-Adoption/Opinion: 20.05.2021
Begründung erhöhter Mengen linearisierter DNA “möglicherweise” gefunden, keine weiteren Details
Studien zur Verbesserung des DNase-Schritts wurden eingeleitet, jedoch keine Berichte oder Ergebnisse an EMA übermittelt
Keine Validierung von Zuliefererangaben, keine hauseigenen Testmethoden oder Spezifikationen für Enzymaktivität eingeführt
Type IB Variation report IB/0055
Einreichung: 28.07.2021
CHMP-Adoption/Opinion: 17.08.2021
Für Standort Andover wird Implementierung von Gegenkontrollen zugelieferter Enzyme behauptet
Wegen fehlender Korrelationsstudie zwischen den Zulieferer-Ergebnissen und den neuen Hersteller-Testmethoden sind diese Kontrollen nutzlos
Zu den fünf neuen Testverfahren liegen weder Beschreibungen, Daten, noch Zahlenwerte für die Spezifikationen vor
Pfizer-BioNTech verweigern Herausgabe der Studien zur DNase-Enzymaufbereitung
Nachweis der Korrelation zwischen DNase-Aktivität und Rest-DNA verlangt
Trotz bestehender Nichterfüllung der Punkte REC #3 & REC #7 wird die Variation zugelassen
Type IB Variation report IB/0106/G
Einreichung: 24.01.2022
CHMP-Adoption/Opinion: 31.03.2022
Beschreibung der Andover-Testmethoden für Zulieferer-Enzyme eingereicht
Weiterhin keine Korrelationsdaten zwischen gemessener DNase-Aktivität und Rest-DNA. Ein Versprechen seitens der Hersteller, diese Daten nun doch zu erheben, wird aber als ausreichend akzeptiert.
Ausser DNase haben die Enzyme am Standort Andover (wegen weniger als 10 “ausdrücklich unterschiedlichen” und vom aktuellen Hersteller bezogenen Chargen) nur vorläufige Spezifikations-Zahlenwerte
Obwohl “mindestens” zehn “ausdrücklich unterschiedliche” DNase-Chargen vom aktuellen Zulieferer in Empfang genommen wurden, wurden bislang nur zwei Chargen (in insgesamt 4 Lieferungen) in der Herstellung verwendet. “Zu wenige” DNase-Chargen werden auch als Begründung für das fehlende Korrelat zwischen Enzymaktivität und Rest-DNA-Mengen herangezogen.
Obwohl die Spezifikation noch “etabliert” wird gibt es einen Zahlenwert, jedoch mit Hinweisen auf zukünftig niedriger ausfallende Aktivitätsmesswerte und das aktuell wegen “Einschätzung und Revision zwecks Optimierung” geschlossene Auftragslabor für die Zulieferervalidierung
Immernoch keinerlei Liefervalidierungsdaten oder Enzymaktivitätsmessungen für die deutschen Standorte Mainz/Rentschler
Die ursprüngliche DNase-Studie wurde abgebrochen, nachdem Prozessveränderungen zu geringeren DNA-Messwerten führten
DNA-Grenzwert wurde wieder überschritten, also informieren die Hersteller über neue DNase-Studien, aber noch keine Einreichung von Berichten oder Daten
Bei Überschreitung der neuen Enzym-Grenzwerte48 soll erst eine kleine Charge aktiven Wirkstoffs hergestellt werden; wenn die Spezifikationen für Chargenfreigabe erfüllt werden, gilt das Enzym trotzdem als akzeptiert
Type II Variation II/0148/G
Einreichung: 10.10.2022
CHMP-Adoption/Opinion: 08.12.2022
Recommendations #3 & #7 erfüllt, 22 Monate nach Markteinführung.
“commercial-scale”-Prozessverbesserungen an allen Wirkstoff-Standorten implementiert, Qualität des Impfstoffs sei nun gesichert (lange nach Ende der Impfkampagne). Der ebenfalls verunreinigte BA.4/BA.5 bivalente Impfstoff bekam am 12.9.2022 EU-Zulassung
Spezifikationen und Testmethoden für Enzyme an den deutschen Standorten eingeführt
Unter Berücksichtigung der neuen Akzeptanzkriterien wurden die Spezifikationen für Andover, die in der vorangegangenen Einreichung angegeben wurden, für DNase und Pyrophosphatase geweitet
Beschreibung der Prozessverbesserungen mit ausreichend historischen Daten eingereicht, dass “keine weiteren Kleinstudien nötig oder vorgesehen sind, um die Korrelation zwischen DNase-I-Aktivität und Rest-DNA zu zeigen”
Die EMA legt einen überraschenden Mangel an Durchsetzungsvermögen an den Tag. Essenziell wichtige Daten zur Beurteilung des zentralen Herstellungsschritts wurden einfach nicht geliefert - nicht lediglich herausgezögert, sondern verweigert, ohne Konsequenz. Pfizer-Biontech durften (und dürfen) ihr völlig unzureichend aufbereitetes Produkt herstellen und ausliefern, während die EMA hinter verschlossener Tür impotent wiederholt, das Fehlen der Daten sei “inakzeptabel”. Parallel wurden die Zielgruppen stetig erweitert und die kritischen Mängel und Verfehlungen nicht öffentlich kommuniziert. Die 1-Milliarde-Grenze für ausgelieferte Impfstoffe war bereits im Juli 202149 überschritten; Stand Juni 2022 sind mehr als 3,5 Milliarden Impfstoffdosen ausgeliefert worden.50
Mit jeder vergangenen Woche und zusätzlichen Prozentpunkten Impfquote stieg die Überfälligkeit dieser Daten, und die Mitschuld der EMA. Es ist, als hätten Pfizer-BioNTech gewusst, dass keine Behörde Konsequenzen ziehen würde, oder könnte, oder wollte? Von Beginn der Prozess-2-Herstellung Juni 202051 bis Oktober 2022 dauerte es, um die Prozessverbesserungen (die keine waren)52 überall einzuführen.
Gemachtes Messen
Interne Dokumente zeigen, dass die DNA tatsächlich Grundzutat ist, die durch inadäquate Herstellungsprozesse nicht ausreichend entfernt wird. Pfizer-BioNTech haben die ursprüngliche Studie zur DNase nie herausgegeben und erst Oktober 2022 einen anderen historischen Datensatz eingereicht, um REC #7 zu erfüllen. Erst dann waren auch die vermeintlichen Prozessverbesserungen und Analysen an allen Wirkstoffs-Standorten eingeführt - aber wie sind diese Mengen DNA53 an den ganzen Kontrollen und Messungen vorbeigekommen?
Die Antwort ist in den von Pfizer-BioNTech während der Herstellung und Freigabe verwendeten Analysemethoden zu finden.
UV-Spektroskopie
DNA-Gehalt der Plasmid-Brühe vor und nach Linearisierung,
RNA-Gehalt nach dem Proteinase-K-Schritt,
RNA-Gehalt während des UFDF-Schrittes vor und nach Verdünnung,
RNA-Gehalt bei Freigabe des Wirkstoffs
qPCR mit “SYBR Green I”-Fluoreszenz
Rest-DNA bei Freigabe des Wirkstoffs
RiboGreen-Fluoreszenz-Assay
RNA-Gehalt während der Chargen-Formulierung
RNA-Gehalt und Umhüllung bei der Chargenfreigabe
UV Spektroskopie misst die Adsorption der Lösung bei 260nm Wellenlänge und kann nicht zwischen RNA und DNA unterscheiden, was allein schon daran erkennbar ist, dass diese Methode sowohl zur Bestimmung des DNA-Gehalts der Plasmidlösung, als auch zur Bestimmung des RNA-Gehalts während der Wirkstoffherstellung verwendet wird.54 War diese übermäßige Erfassung von modRNA und DNA neben den Nanolipid-Verunreinigungen ein weiterer Grund dafür, dass im Laufe der Skalierung der Spektroskopie-Grenzwert des Wirkstoffs von ~1,7 mg/mL auf ~2,25 mg/mL angehoben,55 und ein weiterer Verdünnungsschritt nötig wurde?
Wie sich herausstellt erfasst UV-Spektroskopie bei 260nm das N1-Methylpseudouridin 40% weniger als Uracil.56 Ein “unknown unknown”; eine Sache, von der man nicht weiss, dass man sie nicht weiss. Ob die Messung der RNA an dieser Stelle der Wirkstoffherstellung tatsächlich so verfälschte Werte ergeben hat, und was die Implikationen für die weitere Herstellung und das Endprodukt sind, müssen von pharmazeutisch-behördlicher Seite dringend klargestellt werden.
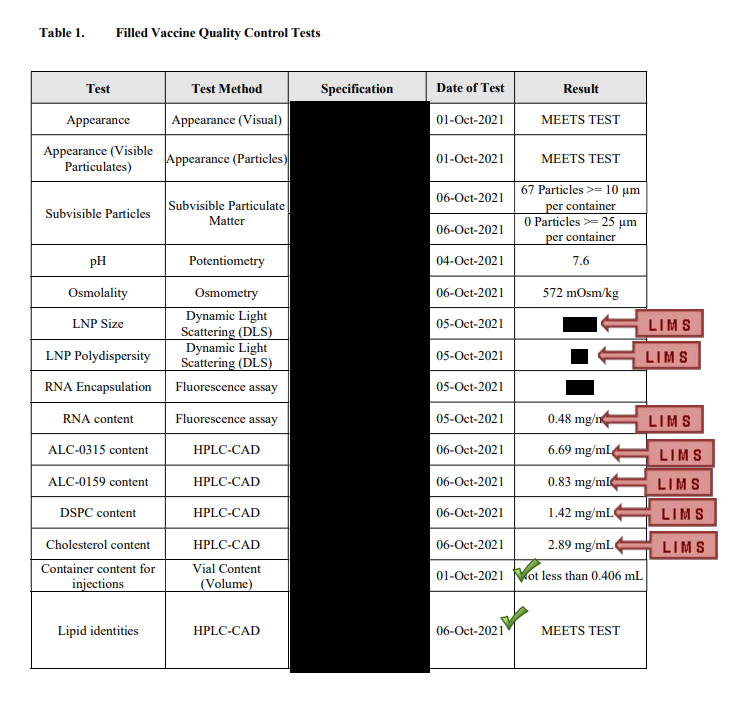
Der SYBR Green I qPCR-Assay zur Bestimmung des DNA-Gehalts wird nur einmal pro Wirkstoff-Chargenfreigabe57 von Pfizer-BioNTech durchgeführt. Meines Wissens wird regulär kein Wirkstoff zur Testung übersandt, nur fertiges Produkt. Im Rahmen der BLA hat die FDA Juni 202158 Wirkstoffproben von drei verschiedenen Chargen verlangt, aber nur die 5’-Cap und Poly-A-Assays wiederholt. Der ermittelte Wert wird vermutlich auf alle Produkt-Chargen übertragen, die aus der entsprechenden Wirkstoff-Charge formuliert werden; zumindestens ist im Gesamtzyklus nur eine Messung vorgesehen. Der qPCR-Assay ist nicht unter den Methodik-Unterlagen59, die per TGA-FOI bekannt sind, was nochmal untermauert, dass dieser Wert von Behörden nie nachgeprüft wurde. Nur die mit LIMS markierten Felder werden einer tatsächlichen Prüfung unterzogen.60 Die neueste Iteration des OMCL-Leitfadens sieht sogar die Prüfung von drei statt vormals vier Eigenschaften vor.61 qPCR ist für die Bestimmung von Rest-DNA nicht geeignet, laut einem Patent von inzwischen Ex-CEO von Moderna, Stephan Bancel;62 Moderna nutzt es nichtsdestotrotz. Stattdessen wird LC/MS/MS vorgeschlagen, eine Methode, die BioNTech in abgeänderter Form für Qualitätskontrolle der Lipide verwendet.
Damit DNA von dem Assay erfasst wird, müssen beide PCR-Primer “treffen”, was kleinere Stücke ausschliesst. Die qPCR-Primer zielen ausserdem auf die T7/Kozak-Sequenz, wo die Bildung der modRNA initiiert wird und somit das höchste RNA:DNA Verhältnis besteht. SYBR Green I bindet an RNA, leuchtet dann aber nicht, weil es nicht PCR-kopiert wird. So bleibt viel DNA ungefärbt, weil die sequenzgleiche und in 100-facher Konzentration enthaltene RNA die PCR-Primer aufsaugt, da ausserdem RNA:DNA über DNA:DNA-Hybridisierung bevorzugt wird.
Die Region mit der höchsten Transkriptionsaktivität als Ziel für die Primer zu nehmen führt auch zu Transkriptionsfehlern, die nicht mehr der Sequenz des Primers entsprechen und somit nicht erkannt werden. Die Methodik hat ausserdem die Schwachstelle, mit manuell verdünnten Fluoreszenzkurven von Referenzmaterial arbeiten zu müssen, was die Vergleichbarkeit der Ergebnisse beeinflussen kann.63
Der RiboGreen-Fluoreszenz-Assay,64 womit in der “drug product”-Herstellung der RNA-Gehalt gemessen wird und als “Fluorescence assay” aufgeführt auf dem Chargen-Freigabebescheid steht, bindet doppelt so stark an DNA, wie an RNA. Jedes erfasste DNA-Molekül gibt doppelt so viel Fluoreszenz ab, wie ein RNA-Molekül. Das Analyseprotokoll sieht aber keine zusätzliche Aufbereitung durch DNase vor.65 Die LNPs werden mit Triton-X aufgelöst, damit die Bindung des Färbstoffs an die Nukleinsäuren nicht behindert wird. Triton-X verfälscht jedoch die UV-Spektroskopie, was wohl die Verwendung von RiboGreen rechtfertigt. Allerdings gibt es auch Farbstoffe mit weniger irreführendem Bindungsverhalten, wie PicoGreen oder AccuGreen.
Das N1-Methylpseudouridin (m1Ψ), dass die Immunogenität der modRNA senkt,66 Dauer und Stärke der Proteinexpression und den Schmelzpunkt erhöht,67 in einem Anteil der Geimpften “off-target” Nonsens-Proteine ausdrückt68 etc. hat weitere Eigenschaften, die einem sauberen und sicheren Produkt nicht unbedingt zuträglich sind. Allein schon, weil die Forschung dazu immer wieder neue Bereiche erschliesst. Die Interaktion der Enzyme T7 Polymerase und DNase mit m1Ψ ist unzureichend studiert, und was bekannt ist, ist nicht gut - die Polymerase bildet statt sauberer Stränge Schleifen, Knoten, und hybride Moleküle, und die Wirkung der DNase auf diese Konstrukte ist wissenschaftliches Neuland, wie auch die Interaktion mit SYBR Green I. Patent_SUN hat anhand Prof. Buckhaults’ Datensätze Kevin McKernan’s experimentelle Feststellung der Verfälschung gemessener Werte durch die Nukleotiden-Modifizierung formularisiert.69
Fazit
Prozess 2 ist inhärent korrupt. Die bekannten zusätzlichen physikalischen Komplexitäten, die bei und durch Modifizierung der RNA entstehen, wurden einfach ignoriert - es ist kein Unterschied zur Synthese “regulärer” mRNA erkennbar. Moderna präsentierte Ende 2022 sogar eine neue, gentechnisch veränderte T7-Polymerase, die weniger “immunologisch aktive” Nebenprodukte erzeugt.70 Die modRNA vereint sich oft genug mit der DNA-Schablone und verhindert dadurch, dass diese enzymatisch zerschnitten wird.71 Die Bereiche des Plasmids, die nicht von der Polymerase abgelesen werden und somit keine entsprechende modRNA haben, sind in viel geringeren Mengen enthalten. UFDF als einzige Wirkstoffreinigung vor der Formulierung in Nanolipide ist archaisch ungeeignet und seit Jahrzehnten nicht mehr dem Industriestandard entsprechend. Das DNA-Konfetti wird zur Streumunition.
Pfizer-BioNTech müssten eigentlich nur eine Polymerase austauschen, um mit der Messung zur Identität der modRNA auch die Menge an DNA zu bestimmen.72 Stattdessen wurde mit Einverständnis sämtlicher Zulassungsbehörden eine kalkulierte Illusion erstellt, indem Eigenschaften verschiedener Messmethoden ausgenutzt werden, um einen willkürlich anmutenden Grenzwert73 einzuhalten. Die Krux der Täuschung liegt darin, dass der Grenzwert für DNA ein Verhältnis74 ist; das qPCR-Ergebnis wird nicht als solches präsentiert75, sondern nur mit dem UV-Spektroskopie RNA-Wert verrechnet. UV-Spektroskopie differenziert nicht zwischen DNA und RNA, sondern zählt beides unabhängig der Länge. qPCR kann DNA unter einer bestimmten Länge gar nicht erkennen, und die Primersequenz ist so platziert, dass der Färbstoff durch RNA aufgesaugt wird und DNA ungefärbt bleibt. Somit entsteht ein Anreiz, DNA unter einer gewissen Länge nicht zu entfernen, um durch das exponentiell höhere Spektroskopie-Ergbnis das Grenzwert-Verhältnis von <330ng DNA/mg RNA einzuhalten. Um auf die nötige Konzentration zu kommen ist ein eigener Verdünnungsschritt76 vorgesehen.
Die zwei Jahre verschleppte Erfüllung der Auflagen ist das Korrelat zum Nachweis überschüssiger Plasmid-DNA in jeder bislang unabhängig untersuchten Ampulle. Somit spielen die nachgereichten Daten, in die wir keinen Einblick erhalten, eine vernachlässigbare Rolle; die vorhandene Evidenz ist schlüssig und wird nur weiter wachsen. Wie lange wird Dhuli et al. der einzige (und durchaus anfechtbare)77 Nachweis von Integration des Impfstoffs sein?
Jedes Covid-PCR-fähige Labor kann Proben aller Art auf SV40 nach McKernan’s Protokoll untersuchen. Die Evidenz spricht dafür, dass diese DNA in jeder Prozess-2-Dosis enthalten ist. Es ist nur eine Frage.. wovon eigentlich?
Vielen Dank an alle, die in kurzer Zeit mit großem Aufwand und Auge fürs Detail gegengelesen haben, fürs Beantworten meiner Fragen und für die umfangreiche Fehlerkorrektur!
Am 12.1.2024 war ich wieder beim Corona-Ausschuss zu Gast und konnte diesen Artikel vorstellen. Die Powerpoint wird noch um den Teil mit Behörden erweitert, das wird etwas größer angelegt.
EMA-Leak Rapporteur's Rolling Review Report Quality - COVID-19 mRNA Vaccine BioNTec.docx
“Linear DNA Template Manufacturing
Cells from the WCB are thawed and the culture is expanded in shake flasks, which are then used to inoculate the fermenter. The culture medium used for expansion and fermentation is a minimal salts/glucose medium that is free of animal-derived components.
Following fermentation, the cells are harvested and chemically lysed to recover the plasmid DNA. After this lysis step, the circular plasmid DNA is purified by ultrafiltration/diafiltration and chromatography.
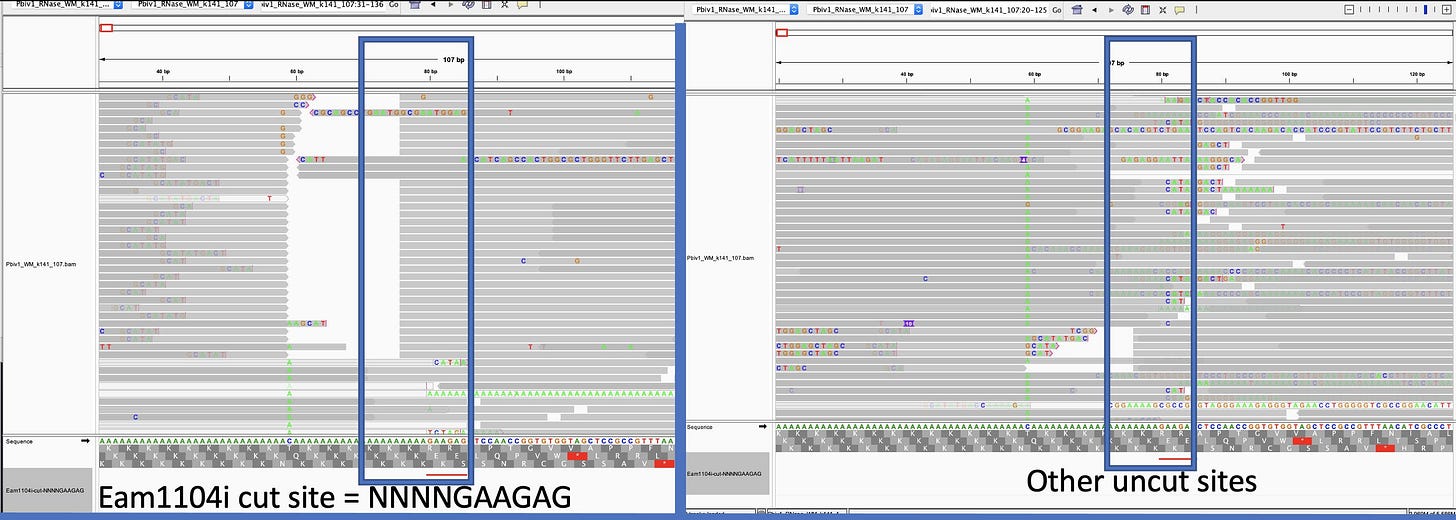
Following purification, the circular plasmid DNA is incubated with a restriction enzyme, Eam1104I or equivalent, in order to linearize the plasmid followed by ultrafiltration/diafiltration. The linear DNA template is filtered and dispensed in a qualified biosafety cabinet in a qualified cleanroom in order to ensure the final linear DNA template is free of any adventitious agent.“
Das Plasmid, von Pfizer-BioNTech eingereicht für Monovalent (EMA-Leak, RRR Report Quality, Seite 24) vs. EPAR-Version Bivalent BA.4/BA.5 vs. von McKernan et al. sequenziert.


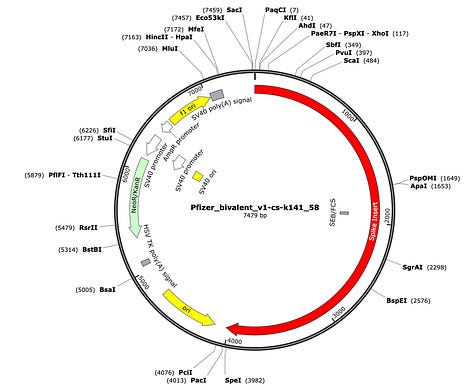
EMA-Leak RRR Report Quality, Seite 23 - “The linear DNA template is produced via plasmid DNA from transformed DH10B Escherichia coli cells.”
EMA-Leak, Rapporteur Report Quality, Seite 31 “After this lysis step, the circular plasmid DNA is purified by ultrafiltration/diafiltration and anion exchange chromatography.“
Failure of the linearization reaction in the Pfizer bivalent vaccine manufacturing process - Nepetalactone Newsletter
DH10B perplexity.ai-Antwort Zu den Modifizierungen gehören verbesserte Aufnahme fremder DNA, Rekombinations-Inkompetenz, entfernte Endonucleasen (die fremde DNA angreifen könnten), stabile Aufnahme großer DNA-Moleküle, und eingebaute zelluläre Marker.
The Complete Genome Sequence of Escherichia coli DH10B: Insights into the Biology of a Laboratory Workhorse
Among the engineered gene replacements were recA1 to improve clone stability by inhibiting the homologous recombination system; endA1, which inactivates the encoded periplasmic DNA-specific endonuclease, thereby enhancing DNA stability during transformation; and a φ80 derivative containing the lacZΔM15 mutation for screening by α-complementation.
Production of the Pfizer BioNTech mRNA jabs - Geoff Pain PhD
“Pfizer jab Manufacturing Process for BNT162b uses modified DH10B Escherichia coli Bacteria cells to grow the DNA used to make their Covid19 Spike Protein encoding mRNA. Interestingly these Bacteria have been designed to "invade" Human Cervix and Breast Cells. Even more interesting is the fact that Chloroquine assists the invasion.”
Siehe Tabelle in Fußnote 1, “Endotoxin ≤20.0 EU/mg”
FDA Dokument Bacterial Endotoxins/Pyrogens “For example, a particular product such as Cyanocobalamin Inj., can have an endotoxin concentration of 10 EU/ml and be considered as in compliance. However, one could question the manufacturer's Water for Injection (WFI) System since the WFI System should have a level of .25 EU/ml.”
S19 M1 Response 2 July 2021, Query 4, Seite 26 “The Bioburden and Endotoxin limits listed in Table 3.2.S.2.2-4 and 3.2.S.2.2-7 are designated as Action limits that would require an action when the limits are exceeded. In Pfizer’s quality system, a test result greater than the action limit requires an investigation and therefore the symbols are accurate as depicted.”
Quelle wie Fußnote 10, “Regarding the action limits for bioburden and endotoxin testing listed in document 3.2.S.2.2 Manufacturing Process Andover, it appears that there are multiple typographical errors (“≤” instead of “>”)..[..]”
Siehe Fußnote 28 & 29. Ein Prozessdokument von Sigma Aldrich zu einer Lösung mit “moderater Viskosität” nutzt ebenfalls eine 300kd-Membran und erwähnt ungefilterte DNA. “Membrane - 300 kD Ultracel® (composite regenerated cellulose) membrane; retains ~10 kD DNA while allowing water, ionic and low molecular weight impurities to pass through”
EMA-Leak, RRR Report Quality, Seite 25 “The plasmid pST4-1525 pre-Master Cell Bank (pre-MCB) was generated by transforming Escherichia coli DH10B competent cells with pST4-1525. A pure culture of transformed cells was produced by growth on selective medium. A single colony isolate was then grown in liquid culture and aliquots were taken and frozen to generate pre-MCB pST4-1525_preMCB_DH10B_20Apr2020.”
Pfizer-interne Bezeichnungen:
PF-07305883 — (nicht-)linearisierte Plasmid-DNA
PF-07305885 — modRNA
PF-07302048 — modRNA-LNP
Durchschnittliche Fragmentlänge mit Oxford Nanopore, RNase-A-behandelter “Pfizer-BioNTech Covid-19 Vaccine” (“Internationale”-Beschriftungsversion von Comirnaty, siehe Fußnote 34). Diese Untersuchung wurde mit Ampure aufbereitet, was Fragmente unter 150bp aussortiert, unterschätzt also stark die Anzahl. Da trotzdm so viele kurze Fragmente sequenziert wurden untermauert, wie massiv die Verunreinigung ist.


“DNase I Demystified”, Thermo Fischer, Twitter-Beitrag
Quelle wie vorangegangene Fußnote “For example, the specific activity of DNase I for ssDNA is about 500 times less than that for dsDNA (4). Activity on RNA-DNA hybrids is <1-2% of that for dsDNA (5).”
Quelle wie vorangegangene Fußnote “DNase I is a sticky enzyme. In some microfuge tubes and 96-well plates we have measured that as much as 50% of the input DNase activity can adhere to the container walls in just 10 minutes! For best results use Ambion's non-stick RNase-free microfuge tubes (Cat #12450) for DNase I digestions.”
Im Rahmen der bekannten Unterlagen sind das Andover und Rentschler. Die anderen Standorte wie Puurs, Polymun und mibe waren Fill/Finish.
Neben der Proteinase K braucht Prozess 1 auch kein Restriktionsenzym, da es nicht linearisiert werden muss. Proteinase K durchläuft eine Schwein-Heparin-Reinigung, ist sonst nicht direkt tierischen Ursprungs; das trifft nur auf den “LB broth”-Nährstoff der Plasmid-Zellkulturen zu. Es enthält hautsächlich Tryptone, ein enzymatisches Verdauungsprodukt von Casein aus Kuhmilch.
EMA-Leak, RRR Report Quality, Seite 187
”Proteinase K used in DS manufacturing and LB broth used in the establishment of the pST4-1525 MCB and WCB are the only materials of animal origin used in the manufacturing of BNT162b2. The applicant has identified contamination of the product by Transmissible Spongiform Encephalopathy (TSE) agents as the main theoretical risk associated with these ingredients.
Proteinase K was manufactured without any human or animal materials but in the purification process a chromatographic column containing heparin of porcine origin was utilized. The LB broth contains casein digest peptone derived from bovine milk fit for human consumption; the origin of the source animals is stated to be Australia and New Zeeland. The TSE/BSE risks for these materials has been discussed under A.2 Adventitious agents safety evaluation and under S.2.3 Control of materials, and is deemed minimal.”
Genaugenommen heisst es keinen direkten Einfluss; die kommerzielle Herstellungsskalierung der Nanolipide erfordert auf Grund von Interaktionen mit Verunreinigungen, die im kleinen Prozess 1 Maßstab nicht vorhanden waren, höhere Konzentrationen RNA. Dieser Mechanismus könnte auch DNA adduktieren, findet aber nach dessen Messung statt.
”A novel mechanism for the loss of mRNA activity in lipid nanoparticle delivery systems”, Nature Communications 2021 Moderna, Inc.
“Nucleic Acid Impurity Reduction in Viral Vaccine Manufacturing” Bioprocess Intl. 2014
Accounting for the Donnan Effect in Diafiltration Optimization for High-Concentration UFDF Applications Bioprocess Intl. 2014
”The Donnan effect influences removal of charged excipients during diafiltration of a high-concentration protein solution. Removal efficiency will be enhanced for excipients with the same charge as the protein and decreased for excipients with a charge opposite the protein. Removal of oppositely charged excipients is analogous to that of partially retained solutes.
Process development scientists should consider excipient removal when designing diafiltration steps. The required number of diavolumes to achieve excipient removal will depend on the extent of the Donnan effect, which is a function of protein concentration, protein charge, and excipient charge.”
Kramberger, Petra et al. “Downstream processing and chromatography based analytical methods for production of vaccines, gene therapy vectors, and bacteriophages.” Human vaccines & immunotherapeutics vol. 11,4 (2015): 1010-21. doi:10.1080/21645515.2015.1009817
”Ultrafiltration, a filtration method based on molecular weight differences between the target compound and impurities can be designed as a very robust method resulting in high virus yields. The scale up of the method to the industrial level is relatively straightforward and if coupled with diafiltration also provides a buffer exchange step. However, precipitation and ultrafiltration alone cannot deliver a product of sufficient purity and need to be combined with other techniques.”
“Best Practices in Nucleic Acid Removal from Vaccine Processes” -Merck Millipore
European Pharmacopeia Free access Covid-19 Seite 148, HepB (CHO) rDNA Impfstoff
“Host-cell- and vector-derived DNA. If mammalian cells are used for production, not more than 10 pg of DNA in the quantity of purified antigen equivalent to a single human dose of vaccine.”
“Theme: Calling and challenging for quantitatively eliminating the underestimation of the DNA contamination in the qPCR” Twitter-Strang || Archiv-Link-Liste
ZIP-Datei wie in Fußnote 1 https://covidvaccinereactions.com/wp-content/uploads/2021/04/EMA-Docs-and-CVR-Edits.zip
Zitat- und Screenshotsammlung als Twitter-Strang
“COVID-19 mRNA Vaccine BioNTech - CHMP Overview and LoQ“ 30.11.2020
Stand April 2023 wurde die EUA für “Pfizer-BioNTech Covid-19 Vaccine” bereits 24 Mal neu ausgestellt. Seitdem sind weitere gefolgt, aber ich habe meinen Artikel noch nicht überarbeitet.
Auf PHMPT ist ein “Development Safety Update Report” einsehbar mit einer Liste aller Marktzulassungen bis April 2021 (Seite 519)
Quelle: vorangehende Fußnote “Target concentrations for the active proteins are defined based on the vendor specifications for activity.“
EMA-Leak COVID-19 mRNA Vaccine BioNTech - CHMP Overview and LoQ Seite 13 Die Formulierung findet in den Unterlagen mehrmals Verwendung “Information on the manufacturing process and process controls for the manufacturing site BNT Mainz & Rentschler is not yet provided. Therefore, the comments below are related only to the Andover site.“
An dieser Stelle erwähnenswert ist die neuseeländische Erstzulassung Februar 2021 mit 58 individuellen Anforderungen. “7. Provide the reassessment of the active substance specification for the DNA template purity and impurities. Due date: July 2021.”
postvac.org-Beitrag || alle vier Dokumente + Korrespondenz || Twitter-Strang || Anfragetext:
Bezeichnend für die Beziehung zwischen Hersteller und Behörde - EMA hatte gerade dieses Aushebeln von überschrittenen Grenzwerten durch Substitution der schlussendlichen (“Release”) Freigabetestung eigentlich verboten. Seite 57 “Priority OCs in quality LoQ”
”1. It is stated that OOS result for in-process controls would trigger an evaluation of the deviation to determine if the batch could be further manufactured. It should be confirmed that OOS results for acceptance criteria will lead to batch rejection.” OOS ist Abkürzung für “out of specification”, also außerhalb der Grenzwerte.
“It is estimated that approximately 1,090,385,631 doses of BNT162b2 were shipped worldwide from the receipt of the first temporary authorisation for emergency supply on 01 December 2020 through 31 July 2021 […]“ July 2021 Pfizer-BioNTech Summary Monthly Safety Report 19736_S0453_M1_smsr-01jul2021-31jul2021.pdf (nur über Oktober 2023 Verzeichnis-Download (1,3Gb ZIP-Datei) verfügbar, der Direkt-Link in der Suchfunktion funktioniert nicht.)
“Bereits am 10.06. behauptet Pfizer in einer Einreichung, Impfstoff “auf Risiko” zu produzieren, was in Anbetracht des erst 16 Tage später stattfindenden “Type C”-Treffens doch relativ auffällig ist.” Betrug unter Aufsicht, Fußnoten 45 & 46
Die von McKernan zuerst sequenzierten Chargen waren Pfizer-BioNTech Bivalent BA4/BA5 mit Herstellungsdatum August 2022 und Zulassungsdatum September 2022.
“Deep sequencing of the Moderna and Pfizer bivalent vaccines identifies contamination of expression vectors designed for plasmid amplification in bacteria” Nepetalactone Newsletter
Siehe Fußnote 30 für die Kalkulationen von Patent-SUN
UV Spektroskopie von Pfizer-BioNTech-Chargen vor (zb. “Pmono1”) und nach (zb. “Pmono1_RNA”) RNase-Behandlung, “UV Spectroscopy” Nepetalactone Newsletter
Siehe Tabelle Fußnote 1 “DNA Concentration (UV260)”
EMA-Leak RRR Report Quality, Seite 97, “The content (RNA concentration) of DS is determined by the measurement of the absorbance of the solution at 260 nm by UV spectroscopy.”
EMA-Leak “M3 20 Nov 2020 FDA Query Responses” Seite 37
“S.2.6.4.3. Composition
The composition of BNT162b2 drug substance remained unchanged with respect to the formulation buffer (10 M HEPES, 0.1 mM EDTA, pH 7.0) during clinical testing and between process 1 and 2. Only the RNA target concentration was increased from 1.7 mg/mL to 2.25 mg/mL already for process 1 to accommodate larger scale LNP manufacturing. No further changes are planned for commercial material.”
stellt einen weiteren Grund klar: die kommerziellen LNP führten während der Formulierung zu Reduzierung der RNA-Integrität und Inhalt. (Siehe Fußnote 27)An dieser Stelle: ihr wichtiger Überblick der bekannten Qualitätsprobleme, und ein brandneuer Artikel, der die LNP-Problematik näher auslegt.
The mRNACalc web server accounts for the hypochromicity of modified nucleosides 2 and enables the accurate quantification of nucleoside-modified mRNA. Esteban Finol, Sarah E. Krul, Sean J. Hoehn, Carlos E. Crespo-Hernández, bioRxiv 2023.07.27.550903; doi: https://doi.org/10.1101/2023.07.27.550903
“More importantly, m1Ψ is hypochromic as compared to uridine at λmax (∆εmax = - 21 %) and, due to the bathochromic shift, m1Ψ absorbs 39.8 % less UV than uridine at 260 nm (Fig. 1c).”
FOI 3390 foi-3390-01.pdf (Lipid HPLC) bis foi-3390-11.pdf (Chargen-Freigabebescheid), Twitter-Strang
EU OCABR Guideline for Pandemic COVID-19 Vaccine (mRNA) alte & neue Versionen || Twitter-Beitrag
Patent US10077439B2 || Twitter-Beiträge von Patent_SUN || Patent tour and freezer clean out Nepetalactone Newsletter
A SYBR Green 1 qPCR assay is used to quantify the residual DNA template fragments after purification of an IVT mRNA solution. however, the primers are aimed at a region of the template with high transcription. how does this primer design influence results?
Weiteres Lesen:


Überraschenderweise validierte die australische Zulassungsbehörde TGA diese Messmethode ca. 8 Monate vor der FDA (Januar 2021 vs. September 2021)
TGA FOI 3390 Document 7 “Quantitation of Total and Percent Encapsulated RNA in Lipid Nanoparticles by RiboGreen Fluorescence Worksheet”
FDA Memorandum “Total RNA content determination for COMIRNATY® final container lots for BLA In-Support Testing.”
“Fluorometry Deep Dive“ Nepetalactone Newsletter Da ich sowieso die Quellen hier übernommen hätte, belasse ich es beim Zitieren des Schlusses.
In Studie BNT162-01 wurden zwei Spike-RBD-Kontrukte an Menschen verabreicht, BNT162b1 mit m1Ψ, BNT162a1 mit U (“unmodifiziert”). BNT162b1 wurde bis auf die höchste Dosierungskohorte mit 60ug vertragen, die nach der ersten Dosis an allen zwölf Teilnehmern beendet wurde, in insgesamt 120 Personen. BNT162a1 wurde nach 2,5 Kohorten oder 30 Geimpften bei einer Dosierung von 3ug wegen schwerer systemischer Nebenwirkungen abgebrochen.
Modifications in an Emergency: The Role of N1-Methylpseudouridine in COVID-19 Vaccines Kellie D. Nance and Jordan L. Meier; ACS Central Science 2021 7 (5), 748-756; DOI: 10.1021/acscentsci.1c00197
Mulroney, T.E., Pöyry, T., Yam-Puc, J.C. et al. N1-methylpseudouridylation of mRNA causes +1 ribosomal frameshifting. Nature 625, 189–194 (2024). https://doi.org/10.1038/s41586-023-06800-3
“Why is DNase I failing” Nepetalactone Newsletter Verschiedene Regionen der linearisierten Plasmid-DNA sind in unterschiedlichen Mengen vorhanden, was auf Schutz gegen Zersetzung durch DNase hindeutet. Dieser Schutz entsteht vermutlich durch “Hybridisierung” der gebildeten modRNA mit der Schablone.
Siehe auch seinen Artikel “DNA-RNA hybrids, R-Loops and nuclease resistance of the mRNA vaccines”
”This brings us into the realm of RNA/DNA hybrids and R-Loops. The RNA:DNA hybridization is favored over DNA:DNA hybridization and this especially true in the case of N1-methylpseudouridine (m1Ψ). Thus the DNA contamination is likely partially hybridized to the modified RNA and this makes it nuclease resistant.”
“How to Eliminate the qPCR underestimation: (step 8) Formularization of the second estimated amount N2” Patent’s Substack
Dousis, A., Ravichandran, K., Hobert, E.M. et al. An engineered T7 RNA polymerase that produces mRNA free of immunostimulatory byproducts. Nat Biotechnol 41, 560–568 (2023). https://doi.org/10.1038/s41587-022-01525-6
Siehe Fußnote 69. Die Kodon-Optimierung ersetzt nicht nur Uracil mit N1-Methylpseudouridin, sondern erhöht auch die G-C-Frequenz. Beides erhöht die “Klebrigkeit” der Nukleinsäure und die Wahrscheinlichkeit von Hybridisierungsereignissen, in denen RNA:DNA in unnatürlichen physikalischen Anordnungen entsteht. Das Thema ist sehr komplex und beim Rausch an der Goldgrube dementsprechend unbeachtet geblieben.
Weiteres Lesen: (japanisches original) DNA-Kontamination durch japanischen Corona-Impfstoff bestätigt
Fluorometry Deep Dive - Nepetalactone Newsletter
”They simply needed to add a 10 minute reverse transcriptase step to provide a RT-qPCR measurement of the RNA. The fact that they intentionally avoided this and moved to a RiboGreen assay known to severely inflate RNA levels when there is DNA contamination is an overt intention to deceive.”
Siehe Fußnote 29 für den 10 pg/Dosis-Grenzwert einer Hepatitis-B-Impfung. Das ist gDNA, nicht linearisierte DNA. Letztere ist ein grundlegend höheres Risiko, ganz zu schweigen von den Bioverteilungs- und Zelleintrittseigenschaften der LNPs.
K. Dhuli, M.C. Medori, C. Micheletti, K. Donato, F. Fioretti, A. Calzoni, A. Praderio, M.G. De Angelis, G. Arabia, S. Cristoni, S. Nodari, M. Bertelli
Presence of viral spike protein and vaccinal spike protein in the blood serum of patients with long-COVID syndrome
Eine kritische Beurteilung: Alley-Oops, Nepetalactone Newsletter





































Excellent work. Thank you. I will need to translate bit by bit but appreciate all the references. Then number of times they changed manufacturers, sources and procedures is absolutely mind boggling. It is really out of control and shows how shoddy this product is and HOW IT HAS NOT IMPROVED by much in 2.5 yrs. There is something there with not only the DNaseI but the other enzymes. They identified and fixed the contaminants in the lipids. How come it didnt happen with the DNase1 etc?
Thanks for linking to my Lipid adduct stuff. More is coming.